機械学習で予測モデルを作るとき、課題のひとつにパラメータのチューニングがあります。
パラメータとはどういう設定値や制限値で機械学習の予測モデルを作るのかを示すものです。
料理に例えると、チャーハンを作る過程が機械学習のアルゴリズムだとすると、どういう具材をどのくらいの量入れるのかがパラメータです。
お米の品種や卵の有無、豚肉か鶏肉か、調味料の種類や量がパラメータになります。チャーハンの良し悪しはこれらパラメータの良し悪しに左右されます。おいしいチャーハンを食べるためには、具材をベストな組み合わせと量で投入する必要があります。

昼食においしいチャーハンを食べたので、チャーハンでたとえました。
話を戻すと、機械学習の決定木の深さであったり、ニューラルネットワークのニューロン数であったり、パラメータは自分で設定する必要があります。機械学習では複数のパラメータを組み合わせて、ベストなレシピを作らねば良い予測モデルは作れません。
SAS Viyaでは各種機械学習アルゴリズムを提供していますが、各機械学習にそれぞれのパラメータが用意されています。料理に例えると、メニューにチャーハンのみならず餃子、ラーメン、寿司、ステーキ、チーズケーキがあるようなものです。シェフ(≒データサイエンティスト)は全てのベストなレシピ(≒パラメータ)を探索せねばならず、労力がいります。
しかし! SAS Viyaには更に便利な機能として、オートチューニングというものが用意されています。
オートチューニングは最も良いパラメータを短い時間で探索してくれる機能です。料理に例えると、究極のチャーハンレシピをViyaが自動的に作ってくれる機能です。夢のようですね。
オートチューニングでは機械学習のパラメータを変えながら複数の予測モデルを作り、最も良い予測モデルのパラメータを探してくれるというものです。決定木だけでもパラメータは10種類以上あるのですが、それらの最良な値をみつけてくれます。
パラメータチューニングを行う際、最も安易な探索方法は各パラメータの全パターンを試すことです。全パターンを試せば、その中から最も良いものはたしかにみつかります。しかし欠点はパラメータチューニングに長い時間がかかってしまい、現実的な手法ではありません。
SAS Viyaのオートチューニングはより賢いパラメータ探索のアルゴリズムを4種類用意しています。
- 遺伝的アルゴリズム(Genetic Algorithm, GA):パラメータを遺伝子と見立てて、淘汰、交叉、突然変異を組み換えすことでパラメータを探索する。
- ラテン超方格サンプリング(Latin HyperCube Sampling, LHS):層別サンプリングの一種で、各パラメータをn個の区間に分割し、区間からランダムに値を取り出してパラメータを探索する。
- ベイズ最適化(Bayesian Optimization):説明変数と予測の間にブラックボックス関数があると仮定し、ブラックボックス関数のパラメータの分布を探索する。
- ランダムサンプリング(Random Sampling):ランダムにパラメータの値を選択して探索する。
探索アルゴリズムを詳しく説明していると終わらないので説明を短くまとめました。SAS Viyaではいずれかのアルゴリズムを利用してオートチューニングを実行することができます。
今回はPythonからSAS Viyaを操作して、オートチューニングを試してみたいと思います。
まずはPython SWATをimportし、CAS Sessionを生成してデータをロードします。
# PythonからCASを操作するためのSWATライブラリをインポート import swat # mysessionという名称のCASセッションを作成 mysession = swat.CAS(host, port, user, password) # mycaslibというCASLIBを作成 add_caslib = mysession.table.addCaslib( name = "mycaslib", session = True, ) # トレーニングデータとしてdm50000_trainをロード dm50000_train = mysession.load_path("Dm50000_train.csv", caslib='mycaslib') # テストデータとしてdm50000_testをロード dm50000_test = mysession.load_path("Dm50000_test.csv", caslib='mycaslib') |
今回はダイレクトメールの送信記録と顧客データを利用します。以下のようなデータです。
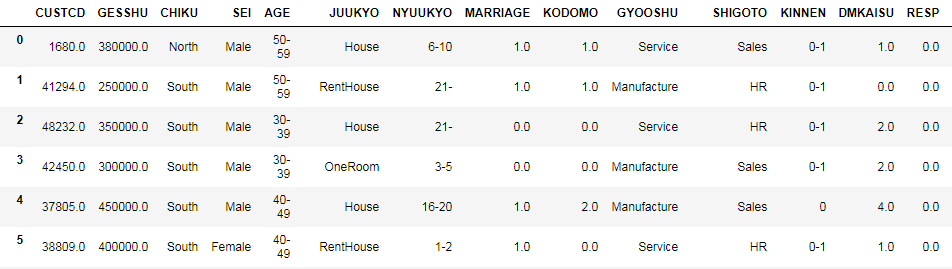
予測のターゲット変数はRESPで、ダイレクトメールに反応したかどうかを格納しています。
-
- 0 = ダイレクトメールに反応しなかった
-
- 1 = ダイレクトメールに反応した
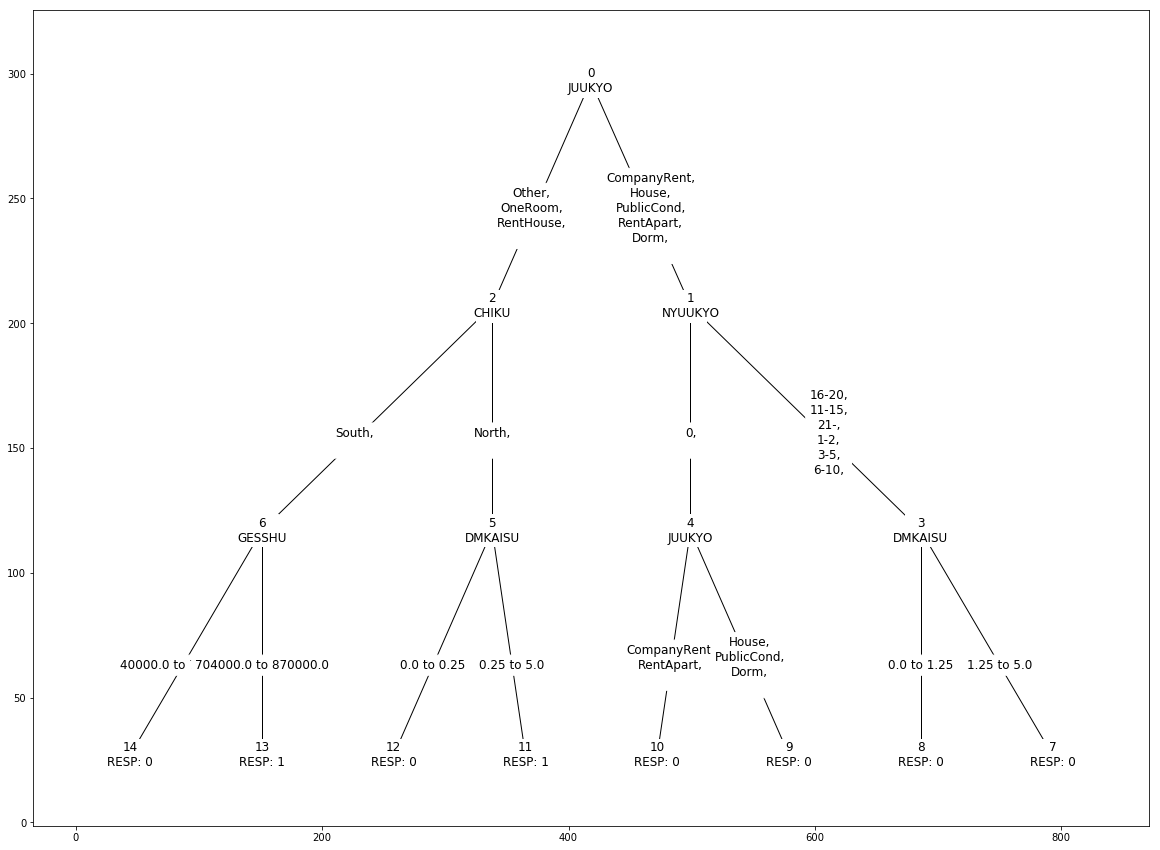
0か1を判定する分類モデルを生成します。機械学習は決定木を使います。 まずはSAS Viyaの決定木を「オートチューニングなし」で使ってみます。
# desicionTree Actionsetをロード mysession.loadactionset("decisionTree") # 決定木で教師あり学習を行い、予測モデルを生成 treeTrain = mysession.decisionTree.dtreeTrain( table={'caslib':'mycaslib', 'name':'DM50000_TRAIN'}, # テーブル名を指定 target="resp", # ターゲット変数はRESP(名義尺度) inputs={'gesshu', 'kodomo', 'dmkaisu', # 使用する説明変数 'chiku', 'sei', 'age', 'juukyo', 'nyuukyo', 'marriage', 'gyooshu', 'shigoto', 'kinnen'}, nominals={'chiku', 'sei', 'age', 'juukyo', 'nyuukyo', 'marriage', 'gyooshu', 'shigoto', 'kinnen', 'resp'}, # ターゲットおよび説明変数のうち、名義尺度を明示 casOut={"caslib":"mycaslib", "name":"dm_tree_scoremodel", "replace":True}, # スコアリングモデルを出力 varImp=True, # 説明変数の重要性を評価 maxLevel=4, # 決定木の深さを最長4に設定 maxBranch=2, # 枝の最大数 leafSize=8 # 葉の最小数 ) |
以下のような決定木が作られます。
2値分類なので、テストデータで予測してROC曲線を引いて精度を評価します。
# 決定木で生成したスコアリングモデルを使用してテストデータをスコアリング treeScore = mysession.decisionTree.dtreeScore( table={'caslib':'mycaslib', 'name':'DM50000_TEST'}, # スコアリング対象にテストデータを指定 modelTable={"caslib":"mycaslib", "name":"dm_tree_scoremodel"}, #スコアリングモデルを指定 casOut={"caslib":"mycaslib", "name":"dm_test_score", "replace":True}, # 結果をdm_test_scoreとして出力 copyVars={"resp"}, # 結果にRESPを追加 assessOneRow=True # 結果に0、1の確率を追加 ) # percentile Actionsetをロード mysession.loadactionset("percentile") # スコアリングされたテストデータをもとに、決定木モデルを評価 treeAssess = mysession.percentile.assess( table={"name":"dm_test_score"}, inputs=[{"name":"_DT_P_ 1"}], response="RESP", event="1", pVar={"_DT_P_ 0"}, pEvent={"0"} ) # ROC曲線を描画 import matplotlib.pyplot as plt plt.figure(figsize=(12,8)) plt.plot(treeAssess.ROCInfo["FPR"], treeAssess.ROCInfo["Sensitivity"], label=treeAssess.ROCInfo["C"][0]) plt.plot([0,1], [0,1], "k--") plt.xlabel("False Positive Rate") plt.ylabel("True Positive Rate") plt.grid(True) plt.legend(loc="best") plt.title("ROC Curve") plt.show() |
最終的に引けたROC曲線は以下のとおりです。
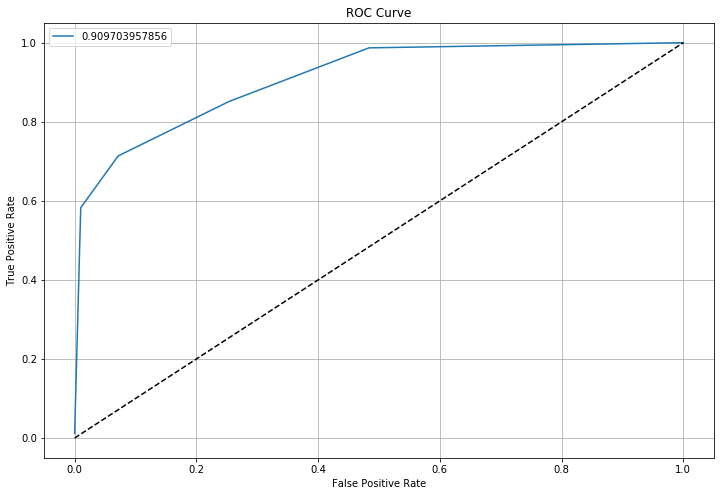
曲線下面積が0.9097...でして、悪くない予測モデルができましたが、まだ改善の余地があるかもしれません。
というわけで、オートチューニングでより良い予測モデルを作ってみましょう。
オートチューニングにはautotuneというアクションセットを利用します。autotuneアクションセットの中には以下のオートチューニングアクションが用意されています。
- 決定木
- ランダムフォレスト
- 勾配ブースティング
- ニューラルネットワーク
- サポートベクターマシン
- ファクタライゼーションマシン
今回は決定木のオートチューニングを行います。
プログラムは以下になります。
# autotuneアクションセットをロード mysession.loadactionset("autotune") # 決定木にオートチューニングを適用 atTrain = mysession.autotune.tuneDecisionTree( trainOptions={ # 学習対象のデータを指定 'table' : {'name':'DM50000_TRAIN'}, # テーブル名を指定 'target' : "resp", # ターゲット変数はRESP(名義尺度) 'inputs' : {'gesshu', 'kodomo', 'dmkaisu', # 使用する説明変数 'chiku', 'sei', 'age', 'juukyo', 'nyuukyo', 'marriage', 'gyooshu', 'shigoto', 'kinnen'}, 'nominals' : {'chiku', 'sei', 'age', 'juukyo', 'nyuukyo', # ターゲットおよび説明変数のうち、名義尺度を明示 'marriage', 'gyooshu', 'shigoto', 'kinnen', 'resp'}, 'casOut' : {"name":"dm_attree_scoremodel", "replace":True} # スコアリングモデルを出力 }, tunerOptions={ # オートチューニングの制限時間やチューニングアルゴリズム等々を指定。 'maxEvals': 49, # 最大検証回数 'maxTime': 3600, # 最長検証時間 'searchMethod': 'LHS', # チューニングアルゴリズム。選択肢:GA, Bayesian, LHS, Random 'seed': 123456, # ランダムシード 'objective': 'RASE' # 目的関数。選択肢:'ASE', 'AUC', 'F05', 'F1', 'GAMMA', 'GINI', 'KS', 'MAE', 'MCE', # 'MCLL', 'MISC', 'MSE', 'MSLE', 'RASE', 'RMAE', 'RMSLE', 'TAU' }, # 探索するハイパーパラメータの制限値 tuningParameters=[{ 'namePath': 'maxLevel', # 決定木の深さを5から8の間に指定 'lowerBound': 5, 'upperBound': 8 }, { 'namePath': 'crit', # 分木方法を以下3つのみに指定 'valueList': {'CHAID', 'CHISQUARE', 'GINI'} }] ) |
オートチューニングではチューニング時のパラメータとして以下を設定することができます。
- trainOptions:対象のデータセット名やターゲット変数、説明変数を指定。
- tunerOptions:オートチューニングの制限時間や制限試行回数、探索アルゴリズム、目的関数を指定。
- tuningParameters:決定木のパラメータのうち、探索対象の値を制限することが可能。
tunerOptionsで制限時間を設定することで、利用者が許容可能な時間内にオートチューニングが完了するように指定できます。時間内に探索できた最良のパラメータが出力となります。
説明変数のうち、既に良い値がわかっている場合はtuningParametersを指定します。例えば決定木の深さは5以上必要ということがわかっている場合、深さ4以下を探索するのは時間の浪費になります。探索対象の値を制限することで、より効率的にパラメータをチューニングすることが可能になります。
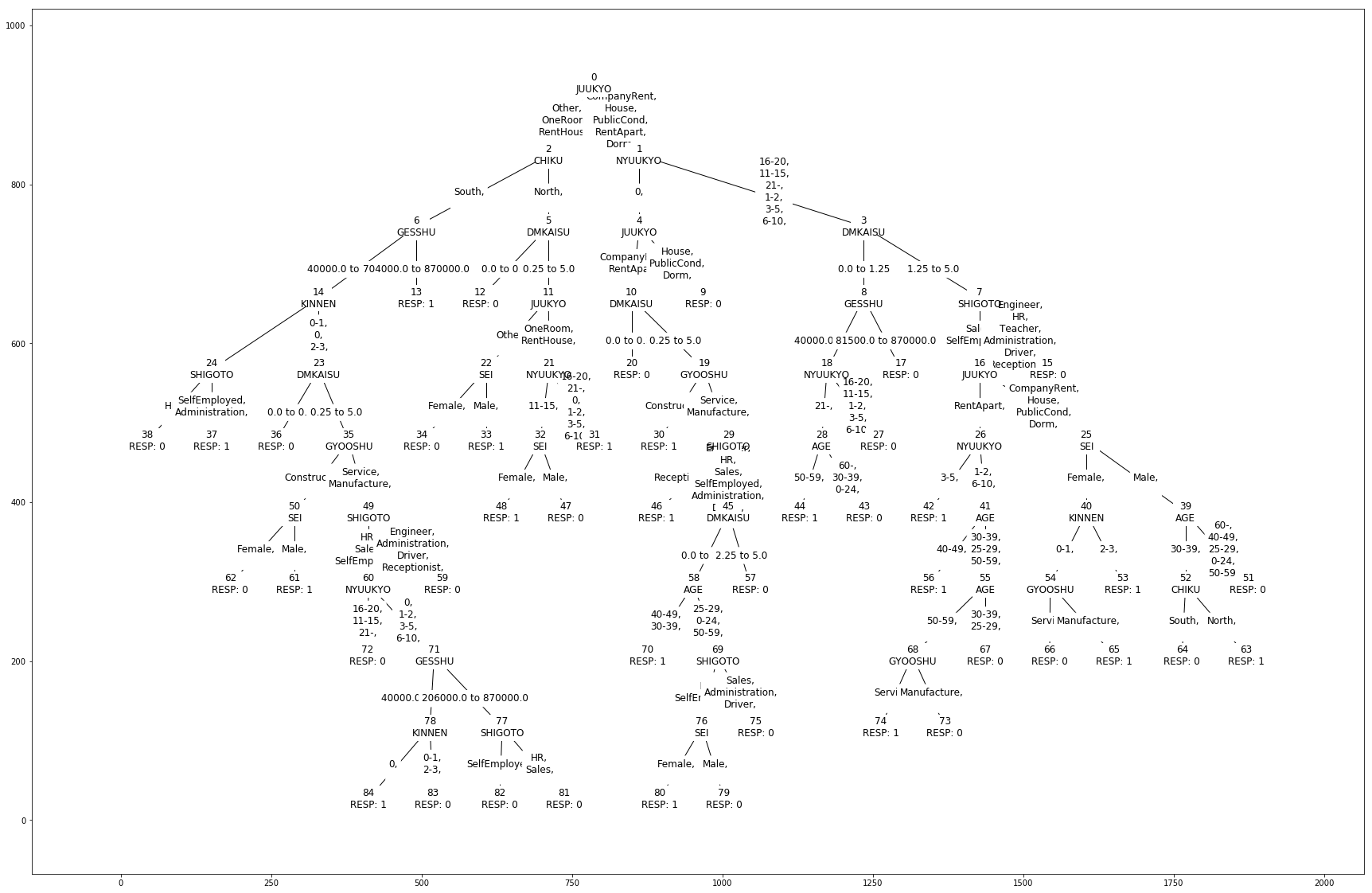
このオートチューニングで生成できた決定木モデルは以下の図になります。
だいぶ複雑で深い決定木が描けたことがわかります。
この予測モデルの精度を評価してみましょう。テストデータをスコアリングし、ROC曲線を描画します。ROC曲線は「オートチューニングなし」と「オートチューニングあり」を重ねて描画し、オートチューニングで精度が改善されていることがわかるようにしています。
# 決定木で生成したスコアリングモデルを使用してテストデータをスコアリング attreeScore = mysession.decisionTree.dtreeScore( table={'caslib':'mycaslib', 'name':'DM50000_TEST'}, # スコアリング対象にテストデータを指定 modelTable={"caslib":"mycaslib", "name":"dm_attree_scoremodel"}, #スコアリングモデルを指定 casOut={"caslib":"mycaslib", "name":"dm_attest_score", "replace":True}, # 結果をdm_test_scoreとして出力 copyVars={"resp"}, # 結果にRESPを追加 assessOneRow=True # 結果に0、1の確率を追加 ) # percentile Actionsetをロード mysession.loadactionset("percentile") # スコアリングされたテストデータをもとに、決定木モデルを評価 attreeAssess = mysession.percentile.assess( table={"name":"dm_attest_score"}, inputs=[{"name":"_DT_P_ 1"}], response="RESP", event="1", pVar={"_DT_P_ 0"}, pEvent={"0"} ) # ROC曲線を描画 import matplotlib.pyplot as plt plt.figure(figsize=(12,8)) plt.plot(treeAssess.ROCInfo["FPR"], treeAssess.ROCInfo["Sensitivity"], label=treeAssess.ROCInfo["C"][0]) plt.plot(attreeAssess.ROCInfo["FPR"], attreeAssess.ROCInfo["Sensitivity"], label=attreeAssess.ROCInfo["C"][0]) plt.plot([0,1], [0,1], "k--") plt.xlabel("False Positive Rate") plt.ylabel("True Positive Rate") plt.grid(True) plt.legend(loc="best") plt.title("ROC Curve") plt.show() |
以下がROC曲線になります。青い線が「オートチューニングなし」、赤い線が「オートチューニングあり」です。
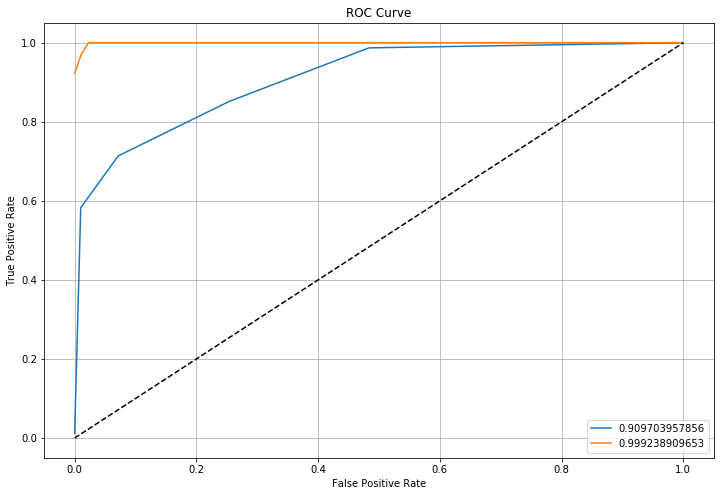
曲線下面積も0.9097...から0.9992...に改善されていることがわかります。
このように、オートチューニングを使うことで自動的に機械学習のパラメータをチューニングし、ベストな予測モデルを効率的に生成することができるようになります。
オートチューニングを利用することで、パラメータチューニングに人的資源を投入する必要がなくなりますし、素早く確実により良い予測モデルを生成することができます。
みなさまもぜひSAS Viyaを使ってオートチューニングを試してみてください。
おまけ
今回作ったプログラムの全文になります。
# 決定木を描画するための関数 # casoutDF: scoring model name from dtreeTrain # saveFig: True for saving a diagram png under /tmp/. default False # size: display size of the tree def drawTree(casoutDF, saveFig=False, size=(20,15)): import networkx as nx from networkx.drawing.nx_agraph import write_dot from networkx.drawing.nx_agraph import graphviz_layout import matplotlib.pyplot as plt import math treeModel = swat.SASDataFrame(mysession.CASTable(casoutDF).head(n=len(mysession.CASTable(casoutDF)))) tree = [] edgeLabel = {} if "_PBName" in treeModel.columns[-3]: liName = [] i = -3 while True: if "_PBNameL" in treeModel.columns[i]: break else: liName.append(treeModel.columns[i]) i -= 1 liName = liName[::-1] for i in range(len(treeModel)): if treeModel.ix[i,"_Parent_"] != -1: if treeModel.ix[i, "_NumChild_"] != 0: pc = (str(int(treeModel.ix[i,"_Parent_"]))+"\n"+treeModel.ix[i,"_ParentName_"], str(int(treeModel.ix[i,"_NodeID_"]))+"\n"+treeModel.ix[i,"_NodeName_"]) tree.append(pc) else: if "_TargetValue_" in treeModel.columns: child = "{0}\n{1}: {2}".format(str(int(treeModel.ix[i,"_NodeID_"])), str(treeModel.ix[i, "_Target_"]), str(int(treeModel.ix[i,"_TargetValue_"]))) pc = (str(int(treeModel.ix[i,"_Parent_"]))+"\n"+treeModel.ix[i,"_ParentName_"], child) tree.append(pc) else: child = "{0}\n{1}: {2}".format(str(int(treeModel.ix[i,"_NodeID_"])), str(treeModel.ix[i, "_Target_"]), str(int(treeModel.ix[i,"_TargetMean_"]))) pc = (str(int(treeModel.ix[i,"_Parent_"]))+"\n"+treeModel.ix[i,"_ParentName_"], child) tree.append(pc) if math.isnan(treeModel.ix[i,"_PBLower0_"]): li = "" for j in liName: if treeModel.ix[i,j] != "": li += treeModel.ix[i,j]+",\n" edgeLabel[pc] = li else: edgeLabel[pc] = "{0} to {1}".format(round(treeModel.ix[i,"_PBLower0_"],2), round(treeModel.ix[i,"_PBUpper0_"],2)) G = nx.DiGraph() G.add_edges_from(tree) plt.figure(figsize=size) pos=graphviz_layout(G, prog='dot') nx.draw_networkx_nodes(G, pos, node_size=1000, node_shape="s", node_color="w") nx.draw_networkx_edges(G, pos, width=1, arrows=False) nx.draw_networkx_edge_labels(G, pos, edge_labels=edgeLabel, rotate=False, label_pos=0.4, font_size=12) nx.draw_networkx_labels(G, pos, font_size=12) if saveFig == True: import uuid file = "/tmp/{0}.png".format(str(uuid.uuid4())) plt.savefig(file) print("saved tree image as {0}".format(file)) plt.show() # PythonからCASを操作するためのSWATライブラリをインポート import swat # 接続先ホスト名、ポート番号、ユーザー名、パスワードを指定 host = "localhost" port = 5570 user = "cas" password = "p@ssw0rd" # mysessionという名称のCASセッションを作成 mysession = swat.CAS(host, port, user, password) # /opt/handson/ディレクトリにmycaslibというCASLIBを作成 add_caslib = mysession.table.addCaslib( dataSource = {"srcType" : "PATH"}, name = "mycaslib", path = "/opt/handson", session = True, ) # トレーニングデータとしてdm50000_trainをロード dm50000_train = mysession.load_path("Dm50000_train.csv", caslib='mycaslib') # テストデータとしてdm50000_testをロード dm50000_test = mysession.load_path("Dm50000_test.csv", caslib='mycaslib') # Dm50000_trainデータセットを全行表示 # セッション名.アクション名(データ名).head(n=行数にDm50000_trainの全行数を指定) mysession.CASTable("Dm50000_train").head(n=len(mysession.CASTable("Dm50000_train"))) # desicionTree Actionsetをロード mysession.loadactionset("decisionTree") # 決定木で教師あり学習を行い、予測モデルを生成 treeTrain = mysession.decisionTree.dtreeTrain( table={'caslib':'mycaslib', 'name':'DM50000_TRAIN'}, # テーブル名を指定 target="resp", # ターゲット変数はRESP(名義尺度) inputs={'gesshu', 'kodomo', 'dmkaisu', # 使用する説明変数 'chiku', 'sei', 'age', 'juukyo', 'nyuukyo', 'marriage', 'gyooshu', 'shigoto', 'kinnen'}, nominals={'chiku', 'sei', 'age', 'juukyo', 'nyuukyo', 'marriage', 'gyooshu', 'shigoto', 'kinnen', 'resp'}, # ターゲットおよび説明変数のうち、名義尺度を明示 casOut={"caslib":"mycaslib", "name":"dm_tree_scoremodel", "replace":True}, # スコアリングモデルを出力 varImp=True, # 説明変数の重要性を評価 maxLevel=4, # 決定木の深さを最長4に設定 maxBranch=2, # 枝の最大数 leafSize=8 # 葉の最小数 ) # 決定木を描画 drawTree("dm_tree_scoremodel", size=(20,15)) # 決定木で生成したスコアリングモデルを使用してテストデータをスコアリング treeScore = mysession.decisionTree.dtreeScore( table={'caslib':'mycaslib', 'name':'DM50000_TEST'}, # スコアリング対象にテストデータを指定 modelTable={"caslib":"mycaslib", "name":"dm_tree_scoremodel"}, #スコアリングモデルを指定 casOut={"caslib":"mycaslib", "name":"dm_test_score", "replace":True}, # 結果をdm_test_scoreとして出力 copyVars={"resp"}, # 結果にRESPを追加 assessOneRow=True # 結果に0、1の確率を追加 ) # percentile Actionsetをロード mysession.loadactionset("percentile") # スコアリングされたテストデータをもとに、決定木モデルを評価 treeAssess = mysession.percentile.assess( table={"name":"dm_test_score"}, inputs=[{"name":"_DT_P_ 1"}], response="RESP", event="1", pVar={"_DT_P_ 0"}, pEvent={"0"} ) # ROC曲線を描画 import matplotlib.pyplot as plt plt.figure(figsize=(12,8)) plt.plot(treeAssess.ROCInfo["FPR"], treeAssess.ROCInfo["Sensitivity"], label=treeAssess.ROCInfo["C"][0]) plt.plot([0,1], [0,1], "k--") plt.xlabel("False Positive Rate") plt.ylabel("True Positive Rate") plt.grid(True) plt.legend(loc="best") plt.title("ROC Curve") plt.show() # autotuneアクションセットをロード mysession.loadactionset("autotune") # 決定木にオートチューニングを適用 atTrain = mysession.autotune.tuneDecisionTree( trainOptions={ # 学習対象のデータを指定 'table' : {'name':'DM50000_TRAIN'}, # テーブル名を指定 'target' : "resp", # ターゲット変数はRESP(名義尺度) 'inputs' : {'gesshu', 'kodomo', 'dmkaisu', # 使用する説明変数 'chiku', 'sei', 'age', 'juukyo', 'nyuukyo', 'marriage', 'gyooshu', 'shigoto', 'kinnen'}, 'nominals' : {'chiku', 'sei', 'age', 'juukyo', 'nyuukyo', # ターゲットおよび説明変数のうち、名義尺度を明示 'marriage', 'gyooshu', 'shigoto', 'kinnen', 'resp'}, 'casOut' : {"name":"dm_attree_scoremodel", "replace":True} # スコアリングモデルを出力 }, tunerOptions={ # オートチューニングの制限時間やチューニングアルゴリズム等々を指定。 'maxEvals': 49, # 最大検証回数 'maxTime': 3600, # 最長検証時間 'searchMethod': 'LHS', # チューニングアルゴリズム。選択肢:GA, Bayesian, LHS, Random 'seed': 123456, # ランダムシード 'objective': 'RASE' # 目的関数。選択肢:'ASE', 'AUC', 'F05', 'F1', 'GAMMA', 'GINI', 'KS', 'MAE', 'MCE', # 'MCLL', 'MISC', 'MSE', 'MSLE', 'RASE', 'RMAE', 'RMSLE', 'TAU' }, # 探索するハイパーパラメータの制限値 tuningParameters=[{ 'namePath': 'maxLevel', # 決定木の深さを5から8の間に指定 'lowerBound': 5, 'upperBound': 8 }, { 'namePath': 'crit', # 分木方法を以下3つのみに指定 'valueList': {'CHAID', 'CHISQUARE', 'GINI'} }] ) # オートチューニングで生成した決定木を描画 drawTree("dm_attree_scoremodel", size=(40,30)) # 決定木で生成したスコアリングモデルを使用してテストデータをスコアリング attreeScore = mysession.decisionTree.dtreeScore( table={'caslib':'mycaslib', 'name':'DM50000_TEST'}, # スコアリング対象にテストデータを指定 modelTable={"caslib":"mycaslib", "name":"dm_attree_scoremodel"}, #スコアリングモデルを指定 casOut={"caslib":"mycaslib", "name":"dm_attest_score", "replace":True}, # 結果をdm_test_scoreとして出力 copyVars={"resp"}, # 結果にRESPを追加 assessOneRow=True # 結果に0、1の確率を追加 ) # percentile Actionsetをロード mysession.loadactionset("percentile") # スコアリングされたテストデータをもとに、決定木モデルを評価 attreeAssess = mysession.percentile.assess( table={"name":"dm_attest_score"}, inputs=[{"name":"_DT_P_ 1"}], response="RESP", event="1", pVar={"_DT_P_ 0"}, pEvent={"0"} ) # ROC曲線を描画 import matplotlib.pyplot as plt plt.figure(figsize=(12,8)) plt.plot(treeAssess.ROCInfo["FPR"], treeAssess.ROCInfo["Sensitivity"], label=treeAssess.ROCInfo["C"][0]) plt.plot(attreeAssess.ROCInfo["FPR"], attreeAssess.ROCInfo["Sensitivity"], label=attreeAssess.ROCInfo["C"][0]) plt.plot([0,1], [0,1], "k--") plt.xlabel("False Positive Rate") plt.ylabel("True Positive Rate") plt.grid(True) plt.legend(loc="best") plt.title("ROC Curve") plt.show() mysession.close() |