はじめに
以前このブログ「グラフ理論入門:ソーシャル・ネットワークの分析例」でもご紹介しましたが。SASは従来からネットワーク分析(グラフ分析)をサポートしています。ネットワーク分析の基本的なことはまず上記のブログをご参照ください。
今回は、プログラミングスキルがあるアプリケーション開発者やデータサイエンティスト向けです。Pythonからネイティブに利用できるSAS Viyaを使用して、ネットワーク分析をする簡単な利用例をご紹介します。
2016夏にリリースされたSAS Viyaは、アナリティクスに必要な全てのアルゴリズムを提供しつつ、かつオープンさを兼ね備えた全く新しいプラットフォームです。これにより、SAS Viyaをアプリケーションにシームレスに組み込むことや、どのようなプログラミング言語からでもアナリティクス・モデルの開発が可能になりました。今回は、SASのパワフルなアナリティクス機能にアクセスするために、そのオープンさがどのように役立つののかにフォーカスします。
前提条件
SAS Viyaは、REST APIにも対応しているため、それを使用しても良いのですが、一般的には、使い慣れたプログラミング言語を使用する方が効率が良いと考えられるため、今回は、データサイエンティストや大学での利用者が多い、Pythonを使用したいと思います。
デモ環境としては、Pythonコードを実行できるだけでなく書式付テキストも付記できる、Webベースのオープンな対話型環境であるJupyter Notebookを使用します。Jupyterをインストールした後に、SAS Scripting Wrapper for Analytics Transfer(SWAT)をインストールする必要があります。このパッケージは、SAS Cloud Analytic Services(CAS)に接続するためのPythonクライアントです。これにより、Pythonから全てのCASアクションを実行することが可能となります。SWATパッケージの情報やJupyter Notebookのサンプルはこちらをごらんください。https://github.com/sassoftware
SAS Cloud Analytic Services(CAS)にアクセスする
SAS Viyaのコアにあるのは、SAS Cloud Analytic Services(CAS: キャス)というアナリティクスの実行エンジンです。"CASアクション"という個々の機能を実行したり、データにアクセスしたりするためには、CASに接続するためのセッションが必要となります。セッションからCASへの接続には、バイナリ接続(非常に大きなデータ転送の場合にはこちらが推奨です)あるいは、HTTP/HTTPS経由のREST API接続のどちらかを使用することができます。今回は、デモンストレーション目的で非常に小さなデータを扱うので、RESTプロトコルを使用します。SAS ViyaとCASのより詳細な情報はこちらのオンラインドキュメントをごらんください。
多くのプログラミングと同様、まずは使用するライブラリの定義からです。Pythonでは、importステートメントを使用します。非常に良く使われるmatplotlibライブラリに加えて、ネットワークをビジュアライズするためのnetworkxも使用します。
from swat import * import numpy as np import pandas as pd import matplotlib.pyplot as plt import matplotlib.colors as colors # package includes utilities for color ranges import matplotlib.cm as cmx import networkx as nx # to render the network graph %matplotlib inline |
『CASに接続してセッションを作成する』という最初のコマンドを実行するために、SWATライブラリをロードしています。パラメータは、環境によってことなります。"s"という変数には、セッションオブジェクトが格納され後で参照するために使用します。
s = CAS('http://sasviya.mycompany.com:8777', 8777, 'myuser', 'mypass') |
アクションセット
CASサーバーは個々のアナリティクス機能であるアクションをアクションセットとして分類します。一つのアクションセットには、複数のアクションが含まれます。アクションには、簡単なデータ操作・管理のための機能から高度なアナリティクスタスクなどまで様々なものがあります。今回のネットワーク分析にには、hyperGroupというアクションが一つだけ含まれている、hyperGroupアクションセットを使用します。アクションセットを利用するためには、以下のように使用を宣言する必要があります。
s.loadactionset('hyperGroup') |
データのロード
アナリティクス・モデリングにはもちろんデータが必要です。サーバー上にすでにあるデータを使用することもできますし、新しいデータをクライアント環境からアップロードすることもできます。データロードの他のサンプルはこちらのサイトでも見ることができます。下の例では、ローカルにあるCSVファイルをサーバーにアップロードし、NETWORK_SAMPLEという名前のテーブルとして格納しています。このテーブルは、FROMとTOという二つの数値型の列を持っています。今回使用するデータはこちらのものを使用しています。 https://en.wikipedia.org/wiki/Zachary%27s_karate_club
inputDataset = s.upload("data/sample_network.csv", casout=dict(name='SAMPLE_NETWORK', promote = True)) |
アナリティクス・モデリングにおいては、データの加工、フィルタ、マージなどももちろん必要になります。ここでは、例として、SASのデータステップコードで新しい列を作成する例を示します。ここで使用しているput関数は、二つの数値変数を変換して、SOURCE、TARGETという二つの文字型の列を作成しています。
sasCode = 'SOURCE = put(FROM,best.); TARGET = put(TO,best.);\n' dataset = inputDataset.datastep(sasCode,casout=dict(name='SAMPLE_NETWORK2', replace = True)) |
データの探索
アナリティクス・モデリングにおける最初のステップは、データを理解することです。例えば、簡単にデータを確認したり、要約統計量を確認したりします。まず、先頭5行を表示してみます。
dataset.fetch(to=5, sastypes=False, format=True) #先頭5行を表示 |
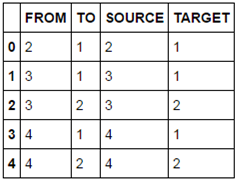
簡単な要約統計量は以下の様になります。
dataset.summary() |
グラフのビジュアライズ
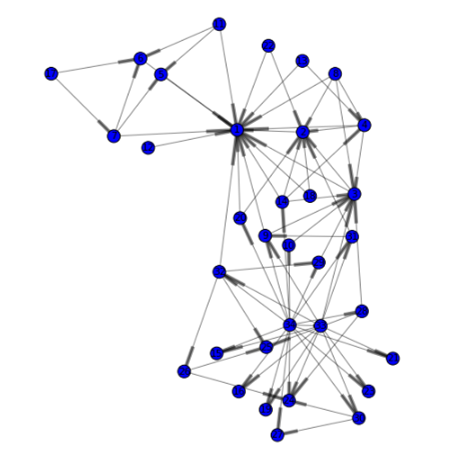
これで準備は整ったので、より詳細にデータを見ていきます。はじめに、基本的なデータ構造と大きさを理解するために、ネットワークをビジュアライズします。先にロードしたhyperGroupアクションを使用し、グラフ描画アルゴリズムである力学モデルを使用して頂点の場所を計算します。hyperGroupはまた、クラスタリングや中心性、コミュニティといったネットワーク評価指標の計算にも利用できます。
s.hyperGroup.hyperGroup( createOut = "NEVER", # これは必要のない出力テーブルの作成を抑制します allGraphs = True, # 孤立していても全てのグラフを処理します inputs = ["SOURCE", "TARGET"], # エッジ(辺)を示す列 table = dataset, # 入力テーブル edges = table(name='edges',replace=True), # エッジ(辺)属性を含む結果テーブル vertices = table(name='nodes',replace=True) # 頂点属性を含む結果テーブル ) renderNetworkGraph(size=10, sizeMultipler=2000) # networkxパッケージを使用してグラフを描画するユーザー定義メソッド |
注)ユーザー定義メソッドであるrenderNetworkGraph()の定義情報は、最下部Appendix A.にあります。
コミュニティの検出
ソーシャルネットワークにおける各ユーザー間の関係性を理解するために、各個人がどのようなコミュニティに属しているのかを分析します。コミュニティ検出、あるいはクラスタリングとは、つながりの強いノード同士をグループ化してネットワークをいくつかのサブ・ネットワークに分解していくプロセスです。hyperGroupアクションでコミュニティ検出をするためには、communityパラメータを指定するだけです。
s.hyperGroup.hyperGroup( createOut = "NEVER", allGraphs = True, community = True, # コミュニティ計算をするためにはtrueを指定 inputs = ["SOURCE", "TARGET"], table = dataset, edges = table(name='edges',replace=True), vertices = table(name='nodes',replace=True) ) |
これで、"nodes"テーブルには新たに、_Community_列が追加され各ノードごとにコミュニティ番号が付与されています。例として、このテーブルの全ての値のディスティンクト・カウントをとったものを表示します。
nodesOut = s.CASTable('nodes') nodesOut.distinct() |
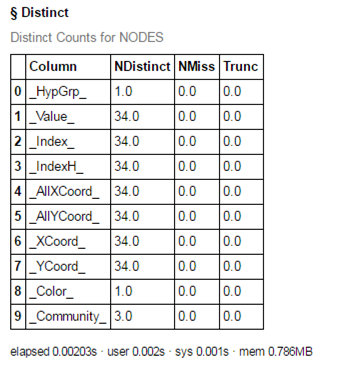
3つのコミュニティが検出されたことがわかります。もし検出されたコミュニティが非常に多い場合には、大きい順にいくつかのコミュニティの情報だけを抽出することも可能です。単純なtopK分析を使用します。
s.simple.topK( aggregator = "N", topK = 2, table = table(name='nodes'), inputs = ["_Community_"], casOut = table(name='topKOut',replace=True) ) topKOut = s.fetch(sortBy=["_Rank_"],to=10, table=table(name='topKOut')) |
表形式の出力ではなくフェッチした行を下で棒グラフの描画に使用するためにPython変数に格納しています。
topKOutFetch = topKOut['Fetch'] ind = np.arange(3) plt.figure(figsize=(8,4)) p1 = plt.bar(ind + 0.2, topKOutFetch._Score_, 0.5, color='orange', alpha=0.75) plt.ylabel('Vertices', fontsize=12) plt.xlabel('Community', fontsize=12) plt.title('# of nodes for the communities') plt.xticks(ind + 0.2, topKOutFetch._Fmtvar_) plt.show() |
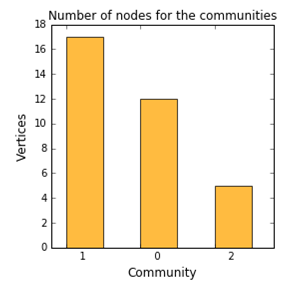
これによると、最も大きなコミュニティは、#1であることがわかります。もちろん各コミュニティの詳細をドリルダウンして見ることもできます。以下の例は#0のコミュニティの内容を表示しています:
nodesOut = s.CASTable('nodes', where="_Community_ EQ 0") nodesOut.fetch(to=5, sastypes=False, format=True) |
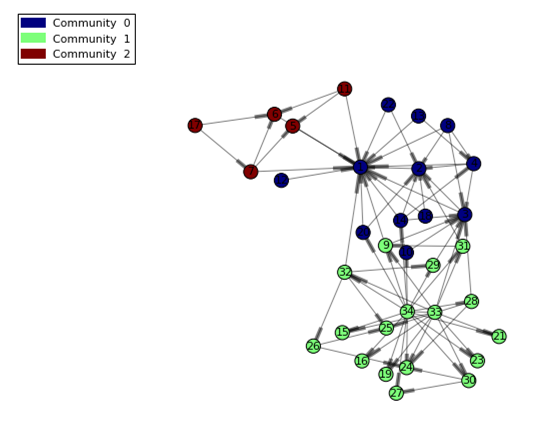
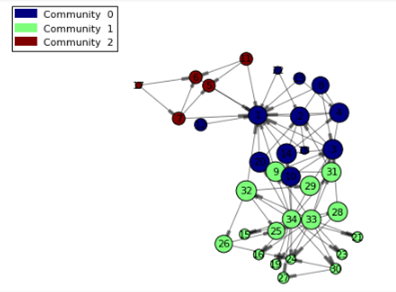
もう一度ネットワークを描いてみます。今回はコミュニティごとに色分けしてみます:
renderNetworkGraph(size=10, colorVar='_Community_', sizeMultipler=2000) |
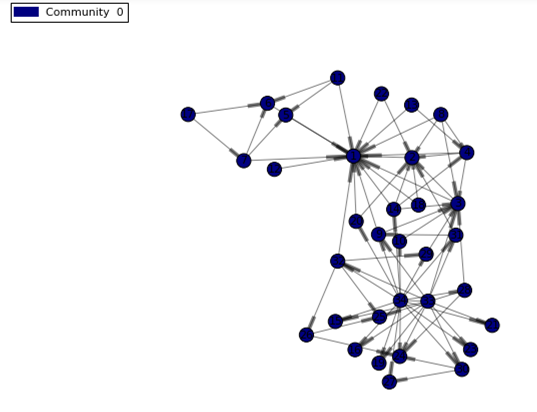
多くの場合、コミュニティの総数はネットワークの大きさやその時々の要件によって調整が必要です。hyperGroupを使用すると小さなグループをどのように大きなグループにマージするかをコントロールすることが可能です。
以下の例ではnCommunityパラメーターを指定することでトータルのコミュニティ数を1にしています。
s.hyperGroup.hyperGroup( createOut = "NEVER", allGraphs = True, community = True, nCommunities = 1, #希望するコミュニティ数 inputs = ["SOURCE", "TARGET"], table = dataset, edges = table(name='edges',replace=True), vertices = table(name='nodes',replace=True) ) renderNetworkGraph(size=10, colorVar='_Community_', sizeMultipler=2000) |
中心性の分析
中心性を分析することでネットワーク上で誰が重要人物かがわかります。重要人物は優れたネットワークを持っておりネットワーク上の他の人に大きな影響を与えます。中心性の概要については、過去のブログ「グラフ理論入門:ソーシャル・ネットワークの分析例」を参照してください。下記の例では、centrality, scaleCentralitiesオプションを使用しています。
s.hyperGroup.hyperGroup( createOut = "NEVER", allGraphs = True, community = True, nCommunities = 5, centrality = True, scaleCentralities = "CENTRAL1", inputs = ["SOURCE", "TARGET"], table = dataset, edges = table(name='edges',replace=True), vertices = table(name='nodes',replace=True) ) |
それでは、ノードのサイズとして中心性指標の一つを使用してネットワークを描画してみましょう。例えば、媒介中心性(Betweenness)は、ある二つのノードの最短経路中に存在する回数を定量化しています。つまり、常にコミュニケーションの仲介役になっているということです。
renderNetworkGraph(size=10, colorVar='_Community_', sizeVar='_Betweenness_') |
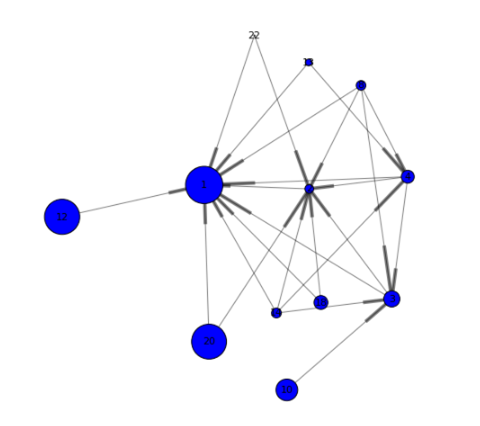
ネットワークのサブセット
今回のネットワークを見ると、コミュニティ#0が重要な役割を持っていることがわかります。このことは、コミュニティの中心性だけでなく、このコミュニティの代表的なノードの高い媒介中心性からも判断することができます。以下のコードによって、このコミュニティだけに絞ったネットワークを描くことができます。
renderNetworkGraph(filterCommunity=0, size=10, sizeVar='_CentroidAngle_', sizeMultipler=5) |
さらに詳細な情報
今回紹介したサンプルの全体のJupyter Notebookはこちらから入手可能です。またAPIリファレンス等は、SAS Viyaのオンラインドキュメントをご参照ください。
今回のサンプルは、単純な2次元の力学モデルを使用したグラフ描画を使用しましたが、実際にはもっと複雑なケースで、次元を追加したいときもあると思います。hyperGroupアクション自体は、三次元にも対応しています。以下の様に、threeDパラメターを使用して3Dに対応した処理を行うことができます。
s.hyperGroup.hyperGroup( createOut = "NEVER", community = True, threeD = True, farAway = 8, inputs = ["SOURCE", "TARGET"], table = dataset, edges = table(name='edges',replace=True), vertices = table(name='nodes',replace=True) ) |
jgraphのようなグラフ描画エンジンを利用することで、対話型ビジュアライゼーションが可能になります。3Dバージョンのサンプルはこちらを参照してください。
まとめ
SAS Viyaは、包括的なアナリティクス機能にセルフサービスでアクセスする環境を提供し、どのようなサイズのデータであっても、またどのような種類のデータであっても扱うことが可能な、一元的に管理可能なアナリティクス・プラットフォームです。パワフルなSASアナリティクス機能を、データサイエンティストは好みの言語-SAS言語, Python, JavaあるいはLua-で利用することが可能です。
Appendix A
def renderNetworkGraph(filterCommunity=-1, size=18, sizeVar='_HypGrp_', colorVar='', sizeMultipler=500, nodes_table='nodes', edges_table='edges'): ''' Build an array of node positions and related colors based on community ''' nodes = s.CASTable(nodes_table) if filterCommunity >= 0: nodes = nodes.query('_Community_ EQ %F' % filterCommunity) nodes = nodes.to_frame() nodePos = {} nodeColor = {} nodeSize = {} communities = [] i = 0 for nodeId in nodes._Value_: nodePos[nodeId] = (nodes._AllXCoord_[i], nodes._AllYCoord_[i]) if colorVar: nodeColor[nodeId] = nodes[colorVar][i] if nodes[colorVar][i] not in communities: communities.append(nodes[colorVar][i]) nodeSize[nodeId] = max(nodes[sizeVar][i],0.1)*sizeMultipler i += 1 communities.sort() # Build a list of source-target tuples edges = s.CASTable(edges_table) if filterCommunity >= 0: edges = edges.query('_SCommunity_ EQ %F AND _TCommunity_ EQ %F' % (filterCommunity, filterCommunity)) edges = edges.to_frame() edgeTuples = [] i = 0 for p in edges._Source_: edgeTuples.append( (edges._Source_[i], edges._Target_[i]) ) i += 1 # Add nodes and edges to the graph plt.figure(figsize=(size,size)) graph = nx.DiGraph() graph.add_edges_from(edgeTuples) # Size mapping getNodeSize=[nodeSize[v] for v in graph] # Color mapping jet = cm = plt.get_cmap('jet') getNodeColor=None if colorVar: getNodeColor=[nodeColor[v] for v in graph] cNorm = colors.Normalize(vmin=min(communities), vmax=max(communities)) scalarMap = cmx.ScalarMappable(norm=cNorm, cmap=jet) # Using a figure here to work-around the fact that networkx doesn't produce a labelled legend f = plt.figure(1) ax = f.add_subplot(1,1,1) for community in communities: ax.plot([0],[0], color=scalarMap.to_rgba(community), label='Community %s' % '{:2.0f}'.format(community),linewidth=10) # Render the graph nx.draw_networkx_nodes(graph, nodePos, node_size=getNodeSize, node_color=getNodeColor, cmap=jet) nx.draw_networkx_edges(graph, nodePos, width=1, alpha=0.5) nx.draw_networkx_labels(graph, nodePos, font_size=11, font_family='sans-serif') if len(communities) > 0: plt.legend(loc='upper left',prop={'size':11}) plt.title('Hartford Drug User Social Network', fontsize=30) plt.axis('off') plt.show() |